Verwendung von Algorithmen des maschinellen Lernens, um tiefe Einblicke in die Zusammensetzung der zellulären Untergruppen zu geben
Zweck
Einführung einer 20-Marker-Testserie, die für die Erfassung auf dem CytoFLEX LX Durchflusszytometer geeignet ist. Die Visualisierung der Daten mit viSNE, FlowSOM und SPADE auf der Cytobank-Plattform wird erforscht. Eine manuelle Gating-Strategie mit der Kaluza Analyse wird gezeigt. Sie wird mit den Untergruppen verglichen, die durch unüberwachtes Clustering mit FlowSOM auf der Cytobank-Plattform identifiziert wurden1,2 Viele dieser Algorithmen sind unbeaufsichtigt. Das reduziert Verzerrungen, die durch manuelles Gating bekannter Subpopulationen eingeführt werden könnte, und ermöglicht es dem Forscher, unerwartete Phänotypen zu identifizieren. Die Reduzierung des Zeitaufwands für eine umfassende Analyse von hochdimensionalen Datensätzen mit Hilfe von Algorithmen des maschinellen Lernens im Vergleich zum manuellen Gating stellt einen zusätzlichen Vorteil dar.
Um die in diesem Anwendungshinweis verwendeten Daten zu generieren, wurden die Blutproben vor der Lyse der roten Blutkörperchen unter Verwendung von Versalyse gemäß dem Standardisierungsverfahren mit einem unten beschriebenen 20-Farben-Antikörper-Cocktail (Tabelle 1) eingefärbt (Teile-Nummer IM3648). Die gefärbten Proben wurden mit einem CytoFLEX LX Durchflusszytometer mit 6 Lasern erfasst. Die unten beschriebene Filterkonfiguration wurde angewandt, um eine optimale Erkennung der einzelnen Farbstoffe zu gewährleisten.
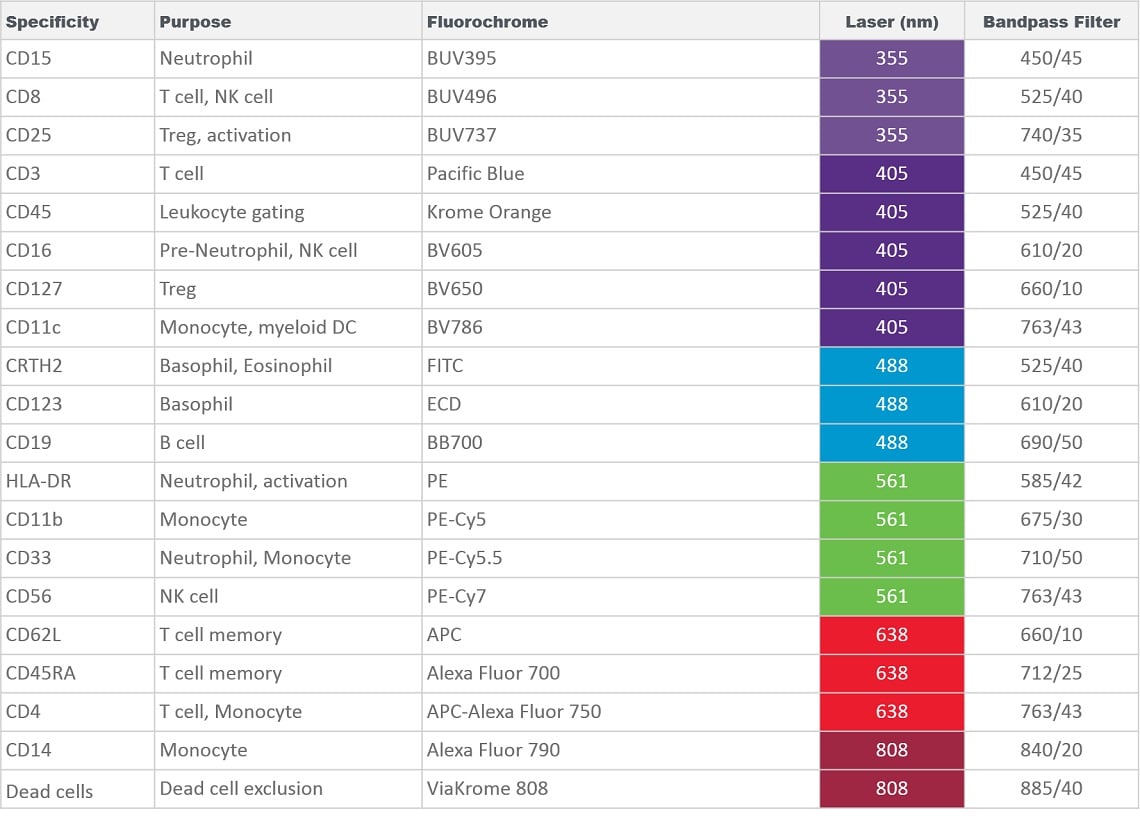
Tabelle 1. Zusammensetzung der Testserie für die Immunophänotypisierung mit 20 Markern mit dem CytoFLEX LX Durchflusszytometer.
Die Kompensation und Datentransformation mittels Logikskalierung wurde mit der Kaluza Analysesoftware durchgeführt. Kompensierte, logisch transformierte Daten wurden mit dem Kaluza Cytobank Plugin in die Cytobank-Plattform exportiert. Die Kaluza Analyse wurde auch für das biaxiale Gating und die manuelle Populationsidentifikation verwendet.
In Vorbereitung auf die maschinelle, lerngestützte Datenanalyse werden Verschmutzungen, Dubletten und oft auch tote Zellen oder andere unerwünschte Ereignisse entfernt. Diese Ereignisse fügen keine Informationen zur Downstream-Analyse hinzu, könnten sich negativ auf die Datenanzeige auswirken und statistische Ergebnisse verfälschen, wenn sie nicht angemessen identifiziert und ausgeschlossen werden. Abhängig von der gewünschten Datenvisualisierung und den wissenschaftlichen Fragestellungen kann es von Nutzen sein, die relevante Population für die weitere Analyse vorzubelegen (Abbildung 1).
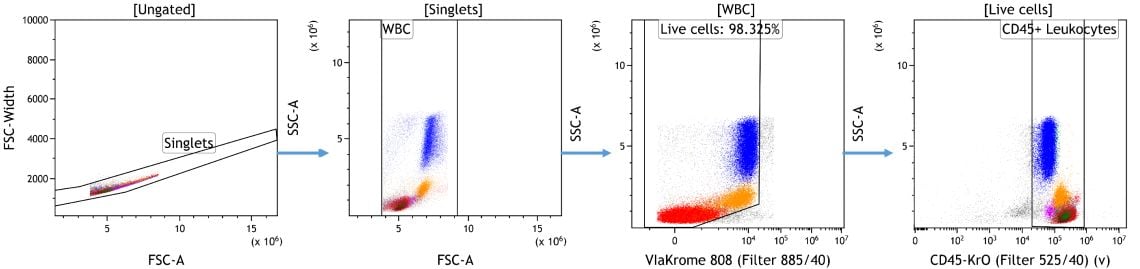
Abbildung 1. Datenbereinigung in der Kaluza-Analyse. Dubletten wurden auf Grundlage der Vorwärtsstreuung des Signalbereichs im Verhältnis zur Höhe ausgeschlossen, gefolgt von einem Gating auf weiße Blutkörperchen auf Grundlage der Merkmale der Vorwärts- und Seitwärtsstreuung. Zellen, die negativ für ViaKrome 808 (Bestellnummer C36628) waren, wurden als lebensfähig identifiziert und anhand der CD45-Expression weiter als Leukozyten klassifiziert. Daten, die mit der Kaluza Analysesoftware analysiert wurden. Plots dienen nur zu illustrativen Zwecken.
Für die manuelle Identifikation von zellulären Untergruppen in Proben aus humanen, venös abgenommenen Blutproben wurde mit Hilfe der Kaluza Analysesoftware eine Gating-Strategie auf der Basis von zuvor veröffentlichtem Wissen über Markerexpressionsmuster erstellt.3
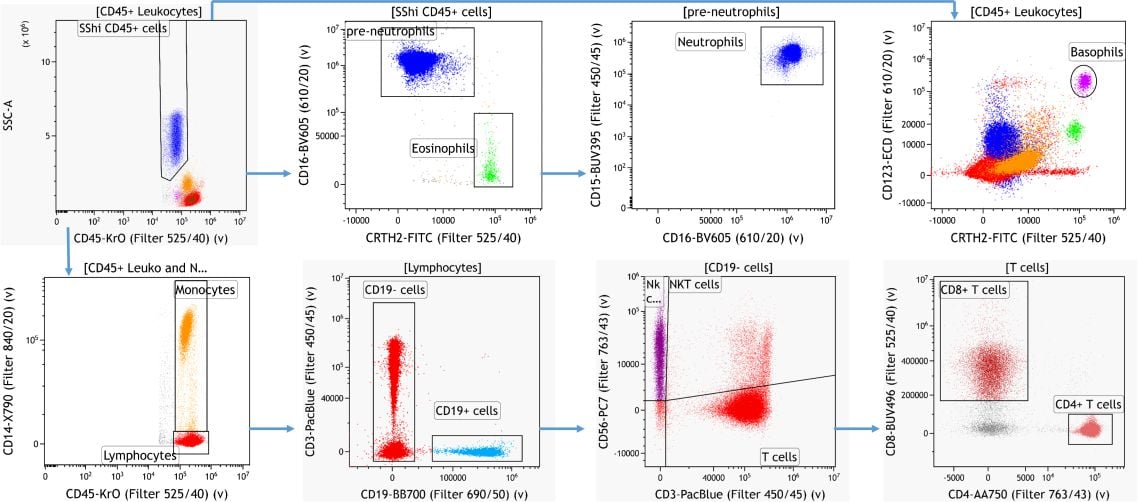 Abbildung 2. Wissensbasierte Identifikation von wichtigen Leukozyten-Untergruppen. Daten, die mit der Kaluza Analysesoftware analysiert wurden. Plots dienen nur zu illustrativen Zwecken.
Abbildung 2. Wissensbasierte Identifikation von wichtigen Leukozyten-Untergruppen. Daten, die mit der Kaluza Analysesoftware analysiert wurden. Plots dienen nur zu illustrativen Zwecken.
Mit Hilfe von Tools des maschinellen Lernens ist es möglich, zelluläre Untergruppen unvoreingenommen und unabhängig von Vorkenntnissen zu identifizieren. Algorithmen zur Reduzierung der Dimensionalität wie viSNE können die Informationen, die in einem hochdimensionalen Datensatz enthalten sind, in einem einzelnen 2D-Plot 1 visualisieren. Cluster-Algorithmen wie FlowSOM können ähnliche Zellen auf Basis von Ähnlichkeiten in der Markerexpression automatisch identifizieren und gruppieren.2
Für die weitere Analyse der Datensätze wurde viSNE zur Reduzierung der Dimensionalität für alle Gating-Marker verwendet, die auch in den manuellen Gating-Schritten verwendet wurden, die in Abbildung 2 dargestellt sind. Dies ermöglicht die Visualisierung der in diesen 11 Markern enthaltenen Informationen (CD45, CRTH2, CD123, CD15, CD14, CD16, CD56, CD3, CD4, CD8, CD19) in einem einzelnen 2D-Plot. viSNE ist eine Methode, hochdimensionale Daten auf zwei Dimensionen zu reduzieren und damit eine schnelle explorative Datenanalyse und Visualisierung komplexer Ergebnisse zu ermöglichen. Bei Zytometriedaten kann dies bei der Kategorisierung von Ereignissen/Zellen in biologische Populationen helfen. Zellen, die sich phänotypisch ähnlich sind, werden nahe beieinander liegen und eine Insel bilden. Ein Konturplot der daraus resultierenden viSNE-Karte ist in Abbildung 3A dargestellt.
Nach der Reduzierung der Dimensionen wurde eine FlowSOM-Analyse durchgeführt, um die Zellen automatisch in 12 sogenannte Metacluster zu clustern. Das Ausführen von FlowSOM auf den populationsbestimmenden Markern und die Anzeige der sich daraus ergebenden Cluster-Daten, die auf der viSNE-Karte überlagert sind, können die Qualitätsbewertung erleichtern. Wenn zur Optimierung der Ergebnisse weitere iterative Anpassungen der Algorithmenlaufeinstellungen erforderlich sind, kann diese Visualisierung dabei helfen, verschiedene Läufe zu vergleichen und einen Ausgangspunkt für die Analyse der Cluster-Daten zu bieten.
Im hier analysierten Datensatz ist eine gute Korrelation zwischen den viSNE-Inseln und den FlowSOM-Metaclustern zu beobachten (Abbildung 3 B). Um den Phänotyp eines jeden Metaclusters schnell zu identifizieren, kann es hilfreich sein, eine Heatmap-Ansicht der FlowSOM-Metacluster durch Clustering zu erstellen (Abbildung 3C).
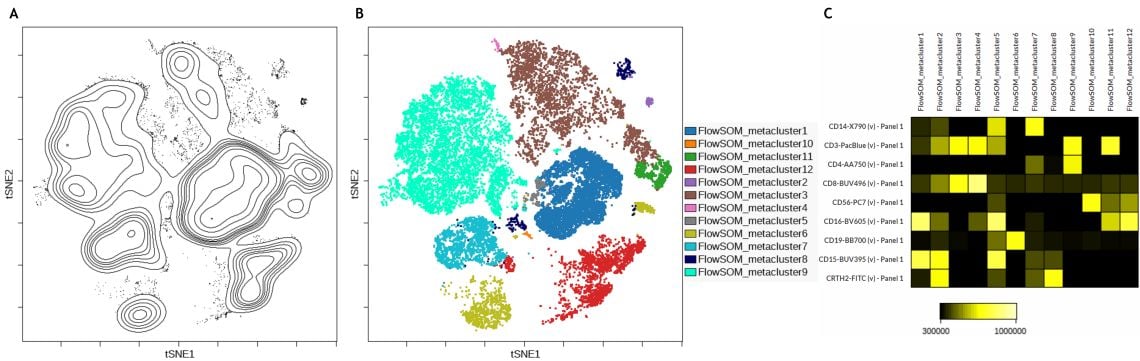 Abbildung 3. Identifikation der Untergruppen mit viSNE, FlowSOM und einer Heatmap-Anzeige. A) Konturplot der viSNE-Karte B) viSNE-Karte mit FlowSOM-Metaclustern als überlagerte Bilddimensionen C) Heatmap-Visualisierung der Markerexpression durch FlowSOM-Metacluster. Die Daten wurden mit Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. viSNE wurde auf 11 populationsbestimmenden Markern von 3 Proben mit 3.000 Iterationen, 30 Perplexität und 0,5 Theta ausgeführt. Die FlowSOM-Einstellungen sind 12 Metacluster und 121 Cluster mit hierarchischem Konsens-Clustering. Plots dienen nur zu illustrativen Zwecken.
Abbildung 3. Identifikation der Untergruppen mit viSNE, FlowSOM und einer Heatmap-Anzeige. A) Konturplot der viSNE-Karte B) viSNE-Karte mit FlowSOM-Metaclustern als überlagerte Bilddimensionen C) Heatmap-Visualisierung der Markerexpression durch FlowSOM-Metacluster. Die Daten wurden mit Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. viSNE wurde auf 11 populationsbestimmenden Markern von 3 Proben mit 3.000 Iterationen, 30 Perplexität und 0,5 Theta ausgeführt. Die FlowSOM-Einstellungen sind 12 Metacluster und 121 Cluster mit hierarchischem Konsens-Clustering. Plots dienen nur zu illustrativen Zwecken.
Die Funktion „Punktplot eingefärbt nach Kanal“, die jedes Ereignis in der viSNE-Karte entsprechend seiner Intensität auf einem Kanal innerhalb des Datensatzes einfärbt, kann verwendet werden, um zu zeigen, warum Punkte in der Karte nahe beieinander liegen oder welche Marker-Expressionsmuster zwischen Ereignissen innerhalb einer viSNE-Insel ähnlich sind. Abbildung 4 zeigt die Markerexpression für CD19, CD4 und CD8 auf der viSNE-Karte und im Vergleich zum FlowSOM-Metaclustering.
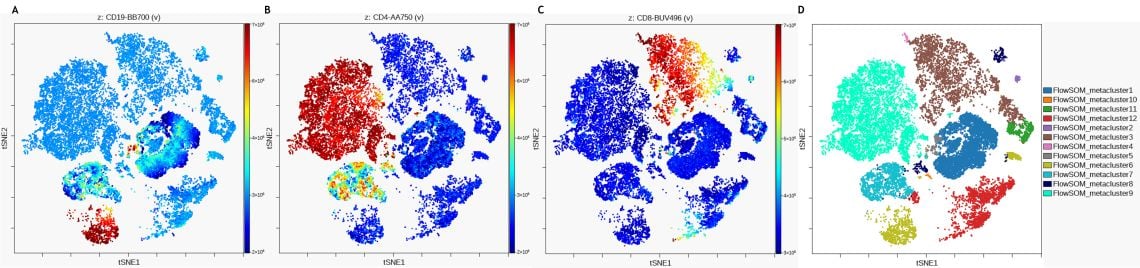
Abbildung 4. Expressionslevel der Subpopulation der Marker auf der viSNE-Karte. A) CD19-Expression B) CD4-Expression C) CD8-Expression D) viSNE-Karte mit FlowSOM-Metaclustern als überlagerte Bilddimensionen. Die Daten wurden mit Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. viSNE wurde mit dem Cytobank Plugin auf 11 populationsbestimmenden Markern von 3 Proben mit 3.000 Iterationen, 30 Perplexität und 0,5 Theta ausgeführt. Die FlowSOM-Einstellungen sind 12 Metacluster und 121 Cluster mit hierarchischem Konsens-Clustering. Plots dienen nur zu illustrativen Zwecken.
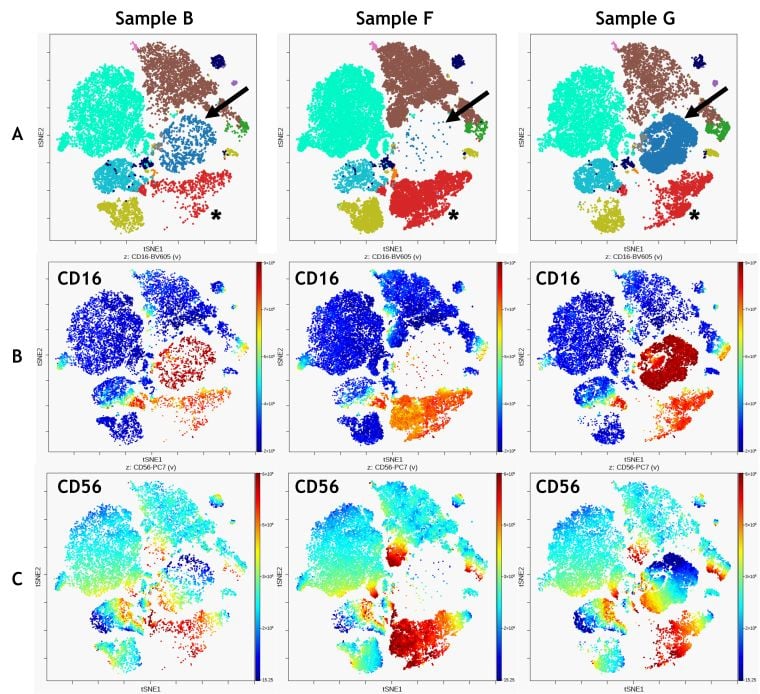
Abbildung 5. Vergleich von 3 Proben. A) FlowSOM-Clustering auf der viSNE-Karte überlagert, Pfeile zeigen Metacluster 1, Sternchen zeigen Metacluster 12 B) CD16-Expression C) CD56-Expression. Die Daten wurden mit Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. viSNE wurde mit dem Cytobank Plugin auf 11 populationsbestimmenden Markern ausgeführt. viSNE wurde auf allen CD45+ Leukozyten von 3 Proben mit 3.000 Iterationen, 30 Perplexität und 0,5 Theta ausgeführt. Die FlowSOM-Einstellungen sind 12 Metacluster und 121 Cluster mit hierarchischem Konsens-Clustering. Die Daten wurden mit der Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. Die weitere Datenanalyse wurde mit auf der Cytobank-Plattform durchgeführt. Plots dienen nur zu illustrativen Zwecken.
Die Kombination von viSNE und FlowSOM ermöglicht qualitative Vergleiche zwischen Proben, die durch die Visualisierung der Expression spezifischer Marker auf der viSNE-Karte verbessert werden können (Abbildung 5). Der Vergleich zeigt, dass die als Metacluster 1 identifizierte CD16+ Population (Abbildung 5 A, blauer Pfeil) in Probe G prominent ist, in den Proben B und F jedoch praktisch nicht vorhanden ist. Außerdem zeigt der Vergleich eine Fülle von hellen CD56-Zellen in Metacluster 12 für Probe F (Abbildung 5 A, rot; Sternchen).
Ein weiterer unüberwachter Algorithmus, der für die Identifikation von Gruppen ähnlicher Zellen verwendet werden kann, ist SPADE. SPADE steht für „Spanning-tree Progression Analysis of Density-normalized Events“ 4. SPADE clustert phänotypisch ähnliche Zellen in einer Rangfolge, die eine hochdurchsatzfähige, multidimensionale Analyse heterogener Proben ermöglicht (Abbildung 6). Es können Blasen hinzugefügt werden, um den verschiedenen; von SPADE gefundenen; berechneten Populationen (Clustern) benutzerdefinierte Populationsschwellenwerte zuzuweisen.
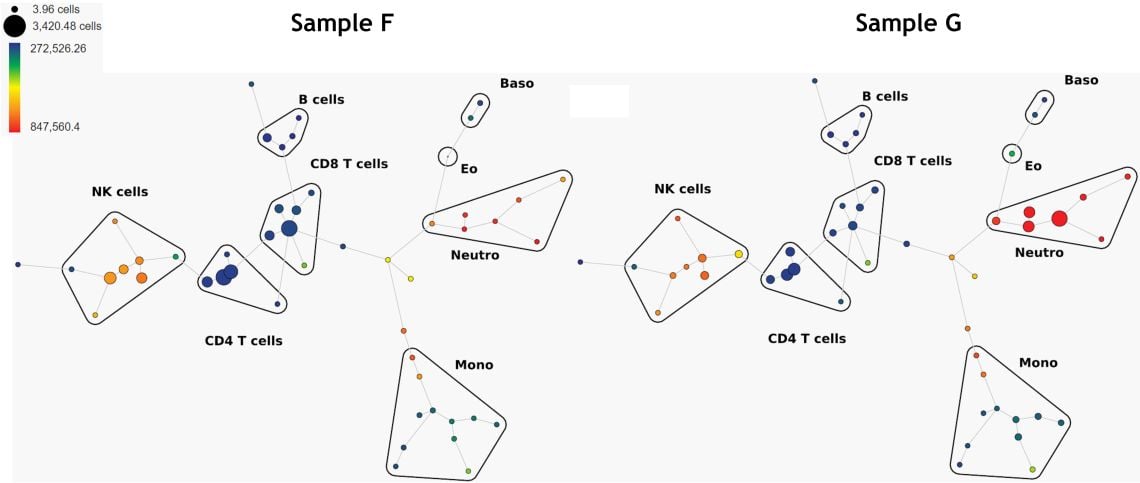
Abbildung 6. Vergleich von 2 Proben, SPADE-Bäume, gefärbt nach CD16-Expression. Die Daten wurden mit der Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. SPADE wurde auf 11 Populationen ausgeführt, die mit Abwärtsproben auf 10 % und 50 Knoten definiert wurden. Plots dienen nur zu illustrativen Zwecken.
Ein tieferes Immunprofil der jeweiligen Leukozytenpopulationen kann durch Erweiterung der manuellen Gating-Strategie erhalten werden. Auch dies basiert im Allgemeinen auf dem bisherigen Wissen über Expressionsmuster. Ein Beispiel für die Untergruppe der CD4+ T-Zellen ist in Abbildung 7 dargestellt.
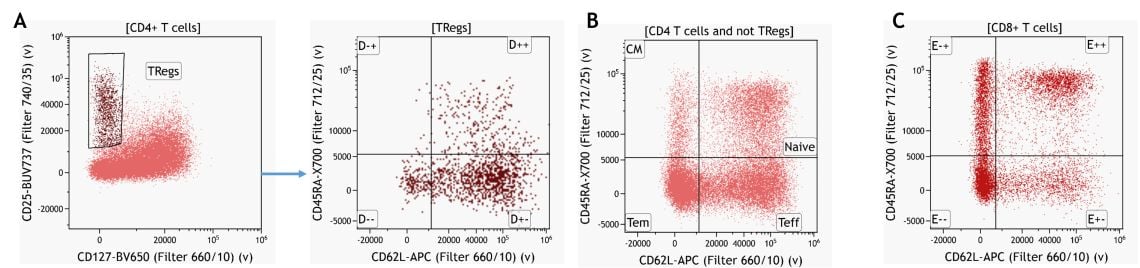
Abbildung 7. Wissensbasierte Identifikation von regulatorischen T-Zellen und deren Untergruppen. (A) Untergruppen der CD4 T-Gedächtniszellen, gated auf CD4+ T-Zellen ohne Tregs (regulatorische T-Zellen) (B) und Untergruppen der CD8+ T-Gedächtniszellen. Daten, die mit der Kaluza Analysesoftware analysiert wurden. Plots dienen nur zu illustrativen Zwecken.
Vergleiche zwischen Proben können durch den Vergleich von Plots oder statistischen Ergebnissen und durch die Verwendung von Overlay-Funktionen oder dem Kaluza-Vergleichsplot (Abbildung 8) durchgeführt werden. Auch dieser Ansatz wird meist von Annahmen über wahrscheinliche Unterschiede geleitet.
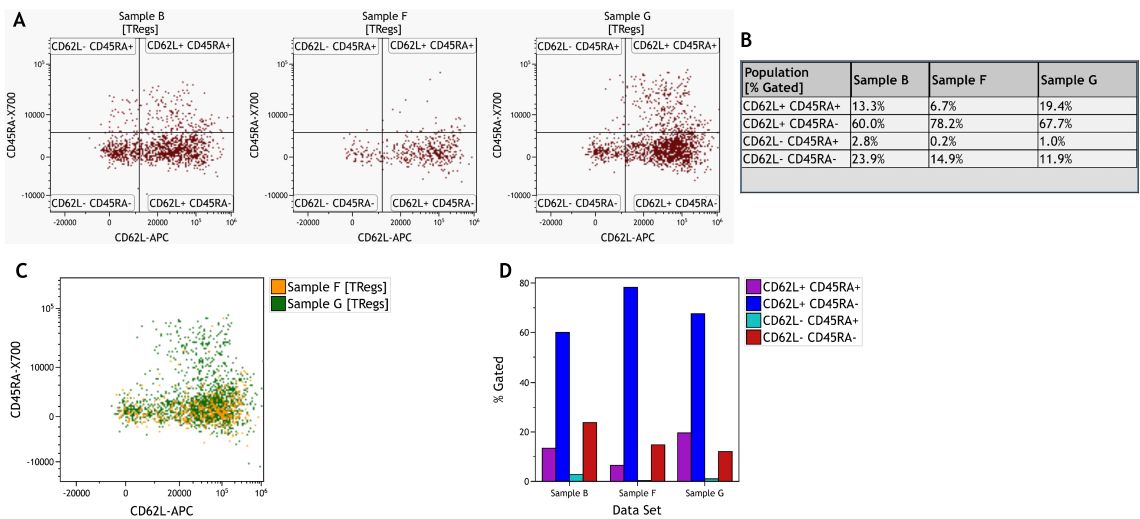
Abbildung 8. Vergleich der Treg-Untergruppe in verschiedenen Proben. A) Einzelne Punktplots pro Probe. B) Informationsblatt mit statistischen Ergebnissen pro Probe und Untergruppe. C) Punktplot-Overlay von zwei Proben. D) Vergleichsplot zur Visualisierung von % Gated pro Untergruppe und Probe. Die Daten wurden mit der Kaluza Analysesoftware analysiert. Plots dienen nur zu illustrativen Zwecken.
Für die unbeaufsichtigte Identifikation von T-Zell-Untergruppen wurde eine viSNE-Analyse mit CD3+ T-Zellen als Eingangspopulation durchgeführt. Abbildung 9 A zeigt die Expression von CD4 und CD8 auf der viSNE-Karte. Nach dem gleichen manuellen Gating-Ansatz wie zuvor (siehe Abbildung 7) wurden unterschiedliche CD45RA- und CD62L-Expressionsmuster mithilfe eines Quadranten-Gates auf Pan-T-Zellen identifiziert und auf der viSNE-Karte visualisiert (Abbildung 9 B). Schließlich wurde ein hierarchisches Konsens-Clustering mit FlowSOM durchgeführt, um 10 Metacluster zu identifizieren (Abbildung 9 C). Sowohl manuelles Gating als auch unbeaufsichtigtes Clustering führen zur Identifikation ähnlicher Populationen.
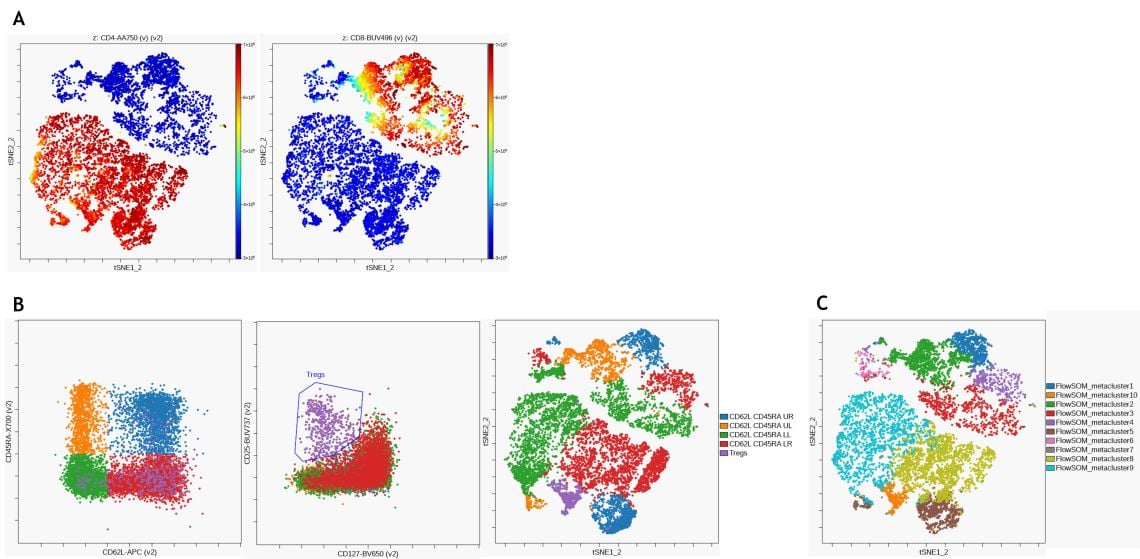
Abbildung 9. Unbeaufsichtigte Analyse von T-Zell-Untergruppen. (A) viSNE wurde auf der Cytobank-Plattform mit 2.000 Iterationen und einer Perplexität von 50 durchgeführt. Die CD4-Expression (links) und CD8-Expression (rechts) werden auf dem daraus resultierenden viSNE-Plot visualisiert (B) CD62L- und CD45RA-Expressionsmuster (links) sowie regulatorische T-Zellen (Mitte) wurden durch manuelles Gating identifiziert und die Populationen auf der viSNE-Karte überlagert. (C) FlowSOM-Clustering wurde mit hierarchischem Clustering auf normalisierten Daten durchgeführt, um 100 Cluster und 10 Metacluster zu erkennen. Metacluster werden auf der viSNE-Karte angezeigt. Die Daten wurden mit der Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. Die weitere Datenanalyse wurde mit auf der Cytobank-Plattform durchgeführt. Plots dienen nur zu illustrativen Zwecken.
Abbildung 10 zeigt den Vergleich der Identifikation der CD8+ Gedächtnisuntergruppe mit manuellem Gating und unbeaufsichtigtem Clustering mit FlowSOM.
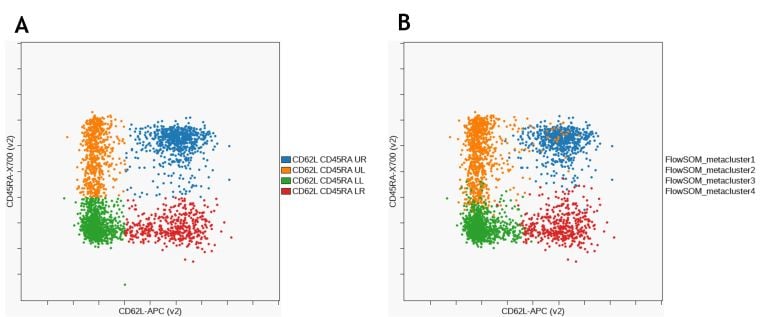
Abbildung 10. Vergleich der Untergruppen der CD8 T-Gedächtniszellen, die durch manuelles Gating identifiziert wurden. (A) und FlowSOM (B). Die Daten wurden mit der Kaluza Analysesoftware kompensiert und logisch transformiert und über das Kaluza Cytobank Plugin auf die Cytobank-Plattform hochgeladen. Die weitere Datenanalyse wurde mit auf der Cytobank-Plattform durchgeführt. Plots dienen nur zu illustrativen Zwecken.
Zusammenfassung
Die Daten von drei Spendern, die mit einer Testserie aus 20-Markern auf einem CytoFLEX LX erfasst wurden, wurden verwendet, um eine manuelle Gating-Strategie zur Identifikation von Leukozyten-Untergruppen sowie eine tiefere Analyse von T-Zell-Untergruppen zu zeigen. Die Verwendung von viSNE zur Visualisierung hochdimensionaler Daten in einer 2D-viSNE-Karte wurde gezeigt und die Verwendung von viSNE und SPADE zum Vergleich von Proben wurde diskutiert. Schließlich wurde die automatische Cluster-Identifikation mit FlowSOM mit den Ergebnissen des manuellen Gatings verglichen. Tools des maschinellen Lernens wie viSNE, FlowSOM und SPADE können bei der Visualisierung von Daten mit hohen Parametern und bei der unvoreingenommenen Identifikation von zellulären Untergruppen hilfreich sein.
Tipps für den Erfolg
Genaue Anleitungen zur Verwendung der Kaluza Analysesoftware finden Sie in der Kaluza IFU C10986. Genaue Anleitungen zur Verwendung der Cytobank-Plattform finden Sie unter support.cytobank.org. Dieses Dokument ersetzt nicht die Gebrauchsinformationen.
Eine ausführlichere Diskussion der hier durchgeführten Analysen findet sich in den Technischen Hinweisen Leveraging the Combined Power of Kaluza and the Cytobank Platform“.
Literaturhinweise
- Amir ED, Davis KL, Tadmor MD, et al. viSNE enables visualization of high dimensional single-cell data and reveals phenotypic heterogeneity of leukemia. Nature Biotechnology. 2013;31(6):545-552. doi:10.1038/nbt.2594.
- Van Gassen S, Callebaut B, Van Helden MJ, et al. FlowSOM: Using self-organizing maps for visualization and interpretation of cytometry data: FlowSOM. Cytometry. 2015;87(7):636-645. doi:10.1002/cyto.a.22625.
- Ortolani C. Antigens. In: Flow Cytometry of Hematological Malignancies. John Wiley & Sons, Ltd; 2011:1-157. doi:10.1002/9781444398069.ch1.
- Qiu P, Simonds EF, Bendall SC, et al. Extracting a cellular hierarchy from high-dimensional cytometry data with SPADE. Nature Biotechnology. 2011;29(10):886-891. doi:10.1038/nbt.1991.
Nur für Forschungszwecke vorgesehen. Nicht für den Einsatz in Diagnoseverfahren.

